Secuenciación de ADN
Para la secuenciación de ADN se emplean dos métodos, el enzimático de terminación de cadena también llamado de Sanger o Desoxi y el de secuenciación automática. En general ambos métodos se desarrollan en tres pasos:
- Realizar la síntesis de nuevos fragmentos de ADN.
- Separar los fragmentos por electroforesis.
- Identificar los nucleótidos para determinar la secuencia.
Para conocer más sobre ambos métodos da clic en los siguientes botones:
- Una molécula pequeña de ADN desnaturalizada, de no más de 500 pb (pares de bases) de tamaño.
- Una polimerasa con la cual se sintetizará la cadena complementaria, por ejemplo la ADNpolimerasa.
- Cebadores más cortos apropiados para esa secuencia.
- Cuatro dNTP ( desoxiribonucleótidos trifosfatos):
- dATP, para la incorporación de Adenina
- dGTP, para la incorporación de Guanina
- dCTP, para la incorporación de Citosina
- dTTP, para la incorporación de Timina
- Cuatro ddNTP (didesoxiribonucleótidos trifosfatos) en pequeñas cantidades, cada uno con un marcador (radioactivo o fluorescente) que permite su localización:
- ddATP, para la incorporación de Adenina: amarillo
- ddGTP, para la incorporación de Guanina: azul
- ddCTP, para la incorporación de Citosina: verde
- ddTTP, para la incorporación de Timina: rojo
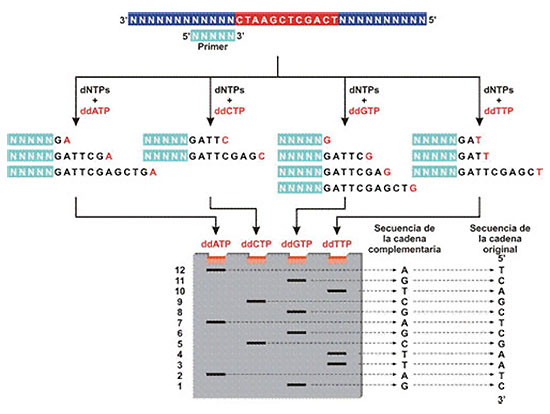
De los cuatro desoxiribonucleótidos (dATP, dCTP, dTTP y dGTP) solo uno se marca radioactivamente. Además se añaden didesoxiribonucleótidos (ddNTPs), estos carecen de un grupo un OH en la desoxirribosa lo que provoca que la polimerasa no pueda leer el siguiente nucleótido y la síntesis se interrumpe siempre en el mismo nucleótido, por tanto, la síntesis de ADN se realiza con tres nucleótidos (dNTPs) y se detiene cada vez que encuentre el mismo ddNTP. Cada segmento de ADN sintetizado termina en el mismo ddNTP.
Cada nucleótido (dNTP) se agrega a un tubo de ensayo diferente, incorporando los requerimientos anteriores para que en cada uno de los tubos se realicen las reacciones que producen una mezcla de ADN de la cadena molde y nuevas cadenas cortas complementarias de diferente longitud que terminan todas en el mismo nucleótido marcadas radioactivamente (todas contienen el cebador empleado en el extremo 5´). La reacción de síntesis se explica porque los dNTP son los sustratos naturales para la síntesis de ADN, contienen el azúcar 2 – desoxirribosa y al utilizar ddNTP que carece de dos grupos hidroxilos (3,2 desoxirribosa) el nucleótido siguiente no puede realizar el enlace, lo que produce que la síntesis se detenga en el sitio en donde el ddNTP específico se une al extremo creciente de la cadena de ADN.
Técnica de separación de ADN basada en la carga eléctrica y el tamaño de las moléculas.
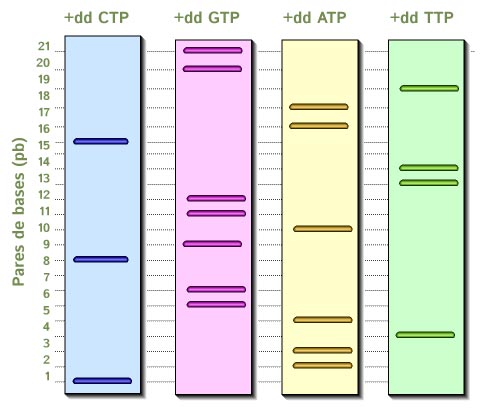
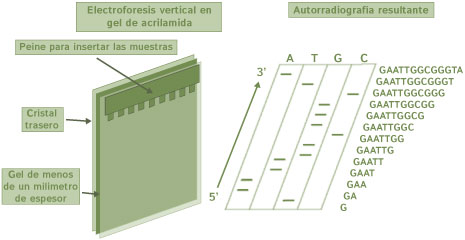
Los numerosos fragmentos de ADN obtenidos se separan de sus moldes y se aíslan por electroforesis en gel de acrilamida vertical de 0.5 mm de espesor y 50 cm de largo, los productos de los cuatro tubos que contienen las mezclas específicas de dNTP se vacían en cuatro carriles diferentes del gel y se deja correr hasta la aparición de bandas que indican que en ese punto está el nucleótido correspondiente al nucleótido empleado.
La lectura se realiza en forma manual mediante la interpretación de la radiografía o bien por medio automatizado que últimamente ha ido desplazando gradualmente el método manual.
Observa la siguiente animación:
Una vez que se completa el corrimiento de la electroforesis se coloca sobre el gel una película fotográfica de autoradiografía. Al autorevelarse la placa, aparecen bandas que indican la posición específica en cada uno de los cuatro carriles, es decir, indican la posición y el tipo de nucleótido (Adenina, Guanina, Citocina o Timina) al que corresponde de tal manera que si la lectura se realiza en dirección 5’ → 3’ la lectura se comienza por las bandas de menor tamaño (extremo 5´) que se encuentran en la base del gel y se continúa hacia las bandas de mayor tamaño (extremo 3´) que se encuentran en la parte superior del gel. La secuenciación se realiza de abajo hacia arriba de la autoradiografía.
Para saber más sobre este tema, te sugerimos revisar la siguiente liga
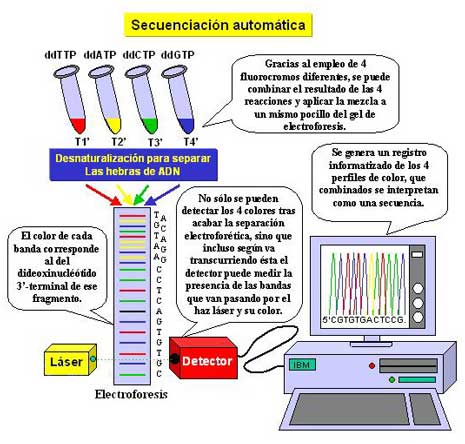
Esta técnica se emplea colores fluorescentes unidos a los nucleótidos en una columna vertical y una computadora lee cada color del nucleótido mediante un rayo láser:
En este método se siguen de manera general los mismos pasos que en el método desoxi con tres diferencias importantes: primero el marcador empleado es fluorescente, segundo la detección del tipo y posición de cada nucleótido (dNTP) se realiza simultáneamente en un solo carril y tercero se emplea una computadora que se programa para analizar la secuencia, de tal forma que la secuenciación se realiza en menor tiempo, con mayor eficiencia y puede ser almacenada en forma digital, sin embargo, los costos de su implementación se elevan.